شناسایی پروتئین به روش مس اسپکترمتری MALDI
ديتا بدست آمده از مس اسپكترومتري MALDI اغلب بصورت تعدادي عدد است كه نماينده ميزان نسبت m/z ذرات پپتيدي هستند. از آنجا كه در اين روش اغلب پروتئينهاي جداسازي شده ( بصورت يك نوع پروتئين و يا تعداد محدودي از پروتئينها) در دستگاه لود مي گردد بنابر اين مقايسه اعداد بدست آمده با مقادير محاسبه شده m/z موجود در Database مي تواند احتمال حضور پروتئين را با درجات اطمينان مختلف نشان دهد.
| Observed mass | Query sequence |
| 768.4705 | K.HVEEVR.K |
| 912.5605 | K.SHEAEVLK.Q |
| 945.5870 | K.KLEAAEER.R |
| 1011.6085 | K.DKHVEEVR.K |
| 1074.6571 | K.DLSLEEIQK.K |
| 1165.6679 | K.AIEENNNFSK.M |
| 1326.8012 | K.ESVPEFPLSPPK.K |
| 1388.8682 | R.ASGQAFELILSPR.S |
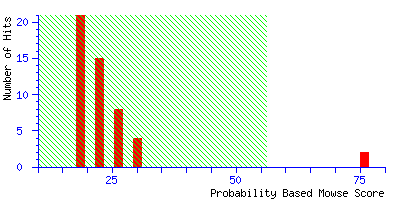
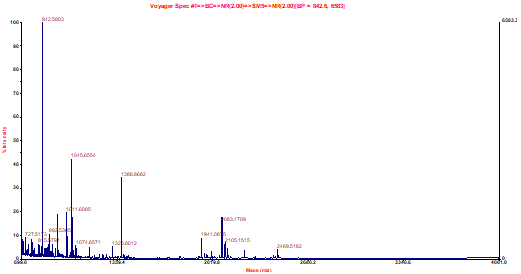
تصوير 1 : تصوير فوق نمايش دهنده دياگرام ارزش احتمالي Mowse (بالا ) و اسپكتراي بدست آمده ( وسط) و پپتيدهاي شناسائي شده جهت پروتئين Statmin در آناليز پروتئين جداشده از 2-D PADE و با روش MALDI-TOF مي باشد (Ref: Sadroddiny et al. IR.J.Asthma, Allergy and Immun, 2011) .
به عبارت ديگر هرچه اعداد بدست آمده با مقدار m/z تئوريك پروتئينهاي Database همخواني بيشتري داشته باشد احتمالا آن پروتئين (يا پروتئينها) در نمونه وجود داشته اند. در تصوير 29 اعداد بدست آمده (جدول) مطابقت بيشتري با پروتئين Stathmin 1 داشته اند. لذا اين پروتئين با درجه اطمينان بيشتري (درجه 75 ) شناسائي شده است.
حضور اسپكترومهاي ديگر ( كه اعداد متناظر آنها در جدول نيامده است) مي تواند نشاندهنده حضور قطعات پپتيدي ديگر و يا آلودگيهاي نمونه باشد.
شناسايي پروتئینها با استفاده از مس اسپکترومتری پشت سر هم ( MS – MS Tandem mass spectrometry , )
مبناي شناسائي پروتئينها در مس اسپكترومتري پشت سر هم همان است كه در روش MALDI توضيح داده شد. با اين تفاوت كه در اين روش علاوه بر داشتن اطلاعات مربوط به نسبت m/z پپتيدها، نسبت m/z اسيدهاي آمينه نيز در دسترس است. اين اطلاعات به ترتيب از آنالايزر اول و دوم بدست مي آيد.
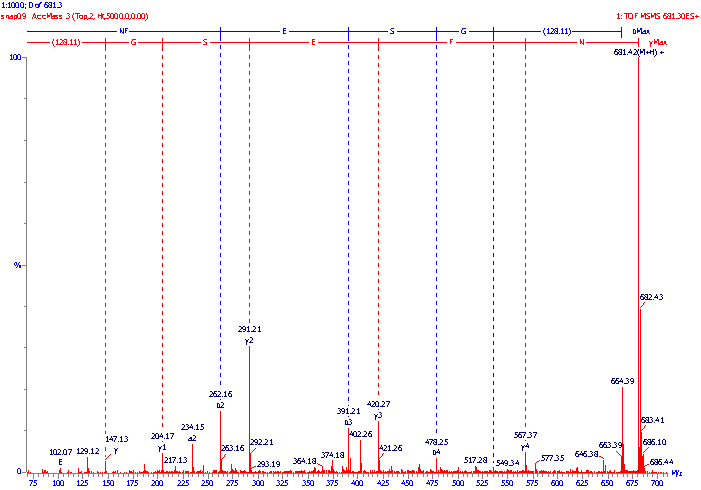
تصوير 2 : توالي يابي پپتيد بوسيله مس اسپكترومتري پي در پي. اسپكتراي يون محصول يا دختر (Daughter or product ion) (Ref: Alison E.Ashcroft, http://www.astbury.leeds.ac.uk/facil/MStut/mstutorial.htm )
از آنجا كه تنها تعداد محدودي از يونهاي ايجاد شده در آنالايزر اول (Precursor ions) پس از تجزيه در واحد تصادم (CID) وارد آنالايزر دوم مي شوند (Daughter or product ions) ، اطلاعات مربوط به اسيدهاي آمينه تنها بخش كوچكي از توالي پروتئين را پوشش مي دهد كه به آن Sequence tag گفته مي شود. با اين حال جستجوي همزمان اطلاعات پپتيدي و اسيدآمينه اي مي تواند شناسائي پروتئين را با درجه اطمينان بالائي انجام دهد.
جدول 1: وزن مولكولي اسيدهاي آمينه مختلف (Ref: Alison E.Ashcroft, http://www.astbury.leeds.ac.uk/facil/MStut/mstutorial.htm )
| Symbol | Structure | Mass (Da) |
| Ala A | -NH.CH.(CH3).CO- | 71.0 |
| Arg R | -NH.CH.[(CH2)3.NH.C(NH).NH2].CO- | 156.1 |
| Asn N | -NH.CH.(CH2CONH2).CO- | 114.0 |
| Asp D | -NH.CH.(CH2COOH).CO- | 115.0 |
| Cys C | -NH.CH.(CH2SH).CO- | 103.0 |
| Gln Q | -NH.CH.(CH2CH2CONH2).CO- | 128.1 |
| Glu E | -NH.CH.(CH2CH2COOH).CO- | 129.0 |
| Gly G | -NH.CH2.CO- | 57.0 |
| His H | -NH.CH.(CH2C3H3N2).CO- | 137.1 |
| Ile I | -NH.CH.[CH.(CH3)CH2.CH3].CO- | 113.1 |
| Leu | -NH.CH.[CH2CH(CH3)2].CO- | 113.1 |
| Lys K | -NH.CH.[(CH2)4NH2].CO- | 128.1 |
| Met M | -NH.CH.[(CH2)2.SCH3].CO- | 131.0 |
| Phe F | -NH.CH.(CH2Ph).CO- | 147.1 |
| Pro P | -NH.(CH2)3.CH.CO- | 97.1 |
| Ser S | -NH.CH.(CH2OH).CO- | 87.0 |
| Thr T | -NH.CH.[CH(OH)CH3).CO- | 101.0 |
| Trp W | -NH.CH.[CH2.C8H6N].CO- | 186.1 |
| Tyr Y | -NH.CH.[(CH2).C6H4.OH].CO- | 163.1 |
| Val V | -NH.CH.[CH(CH3)2].CO- | 99.1 |
